

作者 | 论文团队
编辑 | ScienceAI
想象一下,如果 AI 能拥有一位资深化学家的「化学直觉」—— 不仅能预测药物间的相互作用,更能「看见」分子结构中那些决定其相互作用的微妙联系,甚至揭示出那些沉睡在未知化学空间中的潜在互动,那将是怎样一番景象?
然而,理想与现实之间存在着巨大的鸿沟。一直以来,AI 学习药物相互作用的方式存在一个根本性的瓶颈:它严重依赖于已知的、有标记的药物对数据。这种学习模式导致模型的泛化能力存在先天不足,一旦面对训练数据之外的新药或新组合,其预测精度就会急剧下降,难以推断未知的相互作用规律。
最近,来自于湖南大学的团队在 NeurIPS 2025 接收的一项新研究 ——S²VM,正是在这一背景下提出。其创新地提出了一种有效预测药物相互作用的自监督预训练框架,让 AI 不再仅限于当前有限的药物相互作用数据,而是在广泛的药物空间下探索药物之间潜在关联。

论文标题:Self-supervised Blending Structural Context of Visual Molecules for Robust Drug Interaction Prediction
论文链接: https://neurips.cc/virtual/2025/poster/119726
代码链接: https://github.com/xiaomingaaa/S2VM
1. 引言
联合治疗,即同时使用多种药物,是一种治疗复杂疾病的有效策略。然而,这种方法也带来了其他的问题 —— 潜在的药物 - 药物相互作用(drug-drug interactions,DDIs)可能会改变预期的治疗效果。当患者同时服用多种药物时,这些相互作用可能导致意外的副作用或临床疗效下降。
以往的计算方法 —— 从早期的基于分子结构相似性的模型到后来利用图神经网络(GNN)和知识图谱(KG)的深度学习方法 —— 虽然能够在在已有的药物上得到较为理想的预测效果,但普遍依赖于有限且有偏的已知 DDI 数据。这种监督学习范式限制了模型的「视野」,导致其在应对新药或新组合时泛化能力显著下降。
为突破这一瓶颈,S²VM 提出以大规模未标记药物对为核心的自监督学习框架。该方法不再依赖少量已知交互,而是从约两亿对未探索药物中学习,捕捉分子间的结构特征与潜在关系,从而提升模型对未知药物组合的理解与预测能力。
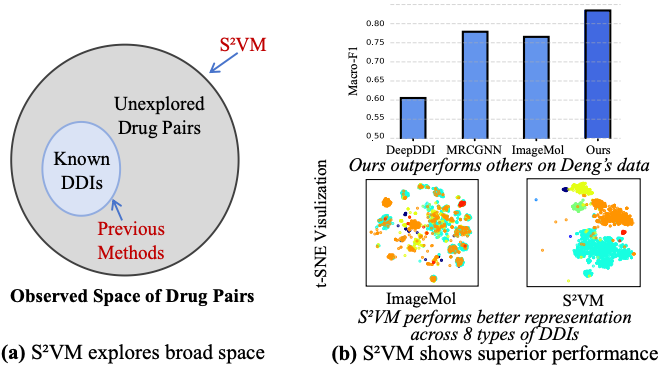
图 1:S²VM 对现有药物的配对空间进行了全面探索。
2.S²VM 方法
S²VM 的核心贡献在于:通过在未标记药物对上的自监督预训练,它成功融合了分子的内在结构表征与外在交互特征,在多种实验场景中实现了 DDI 预测的最新性能。S²VM 方法主要分为四个组件:
在视觉层面融合药物信息:基于局部子结构作为不同分子的共有内在属性这一先验知识,以局部子结构为锚点,对药物对的二维图像进行细粒度视觉融合,从而将分子的内在结构与药物间的外在关系协同编码至一个统一的结构矩阵中。
视觉编解码器:将分子的结构化标记送入一个视觉编码器 - 解码器网络,通过执行编码 - 解码任务来学习并重建其片段间的内在关联,从而捕捉它们深层的语义关系。
重建药物信息:通过解码融合后的统一表示并最小化像素级重建误差,来迫使编码器深入学习药物对的联合视觉结构,最终锻造出一个强大的统一编码器。
下游的 DDI 预测:在下游任务中,预训练的编码器作为一个强大的联合特征提取器,它将输入的药物对信息直接编码为一个高度凝练的全局特征向量,供后续的分类头进行端到端的相互作用预测。

图 2:S²VM 框架包含四个组件:(a) 将药物对融合成统一的输入,对其进行采样并将其混合成结构标记;(b) 将结构标记输入基于视觉的编码器 - 解码器,以模拟分子片段的语义关系;(c) 设置了一个重建操作来恢复输入的药物,以促进药物对的结构融合;(d) 采用预训练的编码器来预测潜在的药物相互作用。
3. 结果
性能评估
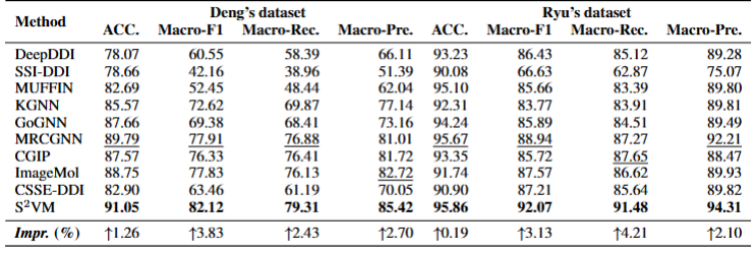
表 1:S²VM 在 Deng 和 Ryu 数据集上的表现。
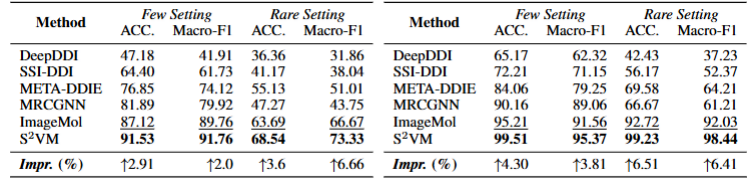
表 2-3:S²VM 在 Deng,Ryu 两个数据集小样本场景下的表现。
DDI 预测模型的真正价值,不仅体现在对大规模已知数据的精准拟合上,更体现在其面对小样本或新药等数据稀疏场景时的预测能力。实验结果显示,S²VM 在各项评测中均展现出更强的性能。在 Deng 和 Ryu 两个标准数据集上,其性能达到了当前最优水平(见表 1)。
更突出的是,在更具挑战性的小样本(Few-shot)预测任务中,其领先优势进一步扩大,展现出强大的鲁棒性(见表 2-3)。这表明,S²VM 通过将药物对视为统一输入进行结构融合,并在海量数据上预先学习精细化的视觉结构表示,不仅提升了常规预测的精度,也显著增强了模型的泛化能力,使其在监督信息极为有限时依然能做出可靠预测。
消融研究
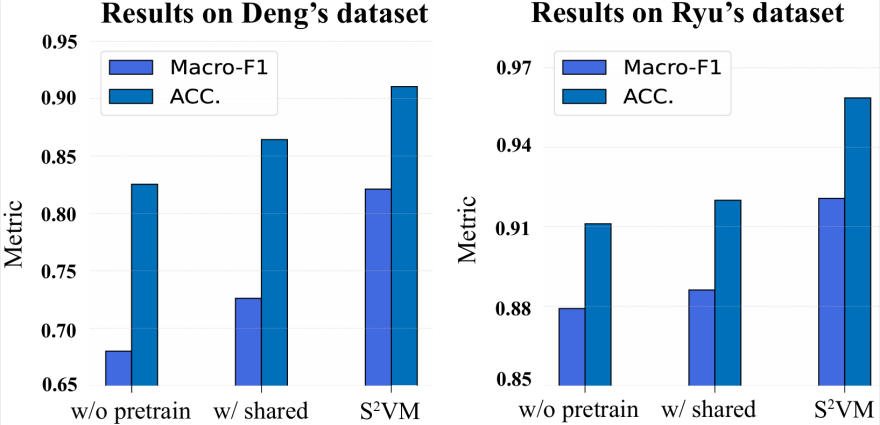
图 2:S²VM 不同变体的结果。
为了客观评估 S²VM 中各项核心设计的贡献,其进行了一系列消融实验,分别测试了移除自监督预训练(w/o pretrain)和替换联合编码为独立编码(w/shared)两种情况。实验结果(见图 2)清晰地表明,在两个标准数据集上,任何核心模块的缺失都会导致模型性能出现显著下降。
这一发现有力地证实了两点:首先,自监督预训练是模型有效提取药物间内外在结构关联的关键;其次,联合编码策略是实现分子视觉信息深度融合的最优路径。因此,S²VM 的卓越性能并非源于单一模块,而是来自于其自监督学习与联合编码策略的紧密结合与协同作用。
有效性和可解释性评估

图 3:S²VM 在 TWOSIDES 数据集上的归纳场景性能。
一个真正强大的 DDI 预测模型,其价值体现在两个层面:一是在新药等数据稀疏场景下的泛化预测能力,二是其预测背后清晰、可信的结构化解释。
S²VM 首先在最具挑战性的新药预测任务中验证了其泛化能力,实验结果(见图 3)表明,在专为评估新药设计的归纳场景(Inductive Scenario)下,S²VM 的表现全面超越了现有方法。这得益于其独特的自监督框架能够从海量的药物对中有效提炼出通用的结构相互作用规律,从而对未知药物做出精准判断。
此外,S²VM 的预测结果更具备坚实的结构化可解释性(见图 4)。在对 DDI 机制的探索中,模型不仅能够精准定位并高亮出导致药物相互作用的关键分子亚结构 —— 例如,在药物 Paroxetine 的案例中,模型成功识别出了文献报道的已知酶抑制剂片段(如 1,3-Benzodioxole),而且进一步的量化评估也证实,模型关注的重点区域与生物学先验知识高度吻合。
卓越泛化能力与深度可解释性的协同作用,使得 S²VM 不仅能进行精准预测,更能提供可靠的机制洞察,使其成为一个稳健可靠的计算工具,能够支持并深化对药物相互作用机制的研究。

图 4:S²VM 的结构化可解释性。
结语
当前药物研发领域坐拥海量分子数据,但如何将其转化为有效的生物学洞察仍是巨大挑战。S²VM 开辟了一条将数亿级未标记药物对直接转化为强大预测能力的有效路径,成功弥合了原始数据与高层知识之间的鸿沟。它使得研究范式从被动等待实验数据的积累,向主动、大规模地从数据中直接发现知识的转变成为可能,从而为根本性加速新药的发现进程铺平了道路。
参考资料:
[1] Ma, Tengfei, et al. "Self-supervised Blending Structural Context of Visual Molecules for Robust Drug Interaction Prediction." The Thirty-Ninth Annual Conference on Neural Information Processing Systems. 2025.
[2] https://github.com/xiaomingaaa/S2VM
人工智能 × [ 生物 神经科学 数学 物理 化学 材料 ]










