面对多模态、跨尺度、强约束的科研问题,通用 LLM 正从“工具”升级为“合作者”。
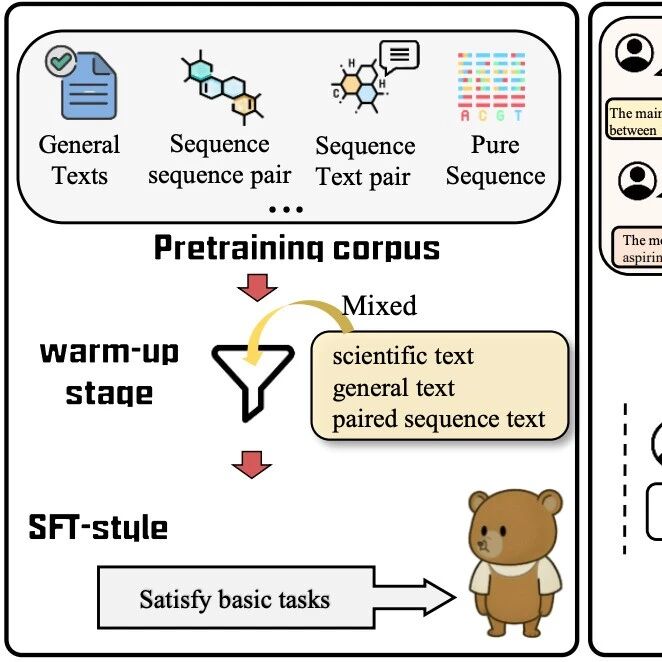
我们带来了一款为科学数据“读—思—设”而生的统一科学基座模型:以更完整的科学数据基座、更系统的训练日程与更可验证的推理机制,直击科学计算与设计痛点。它有三点最值得关注——覆盖更广:统一 I/O 与“任务分组奖励”让单一骨干跨化学/生命/材料等多领域;可验证性更强:从数据标注到思维链再到 RL 奖励全面“科学化”,强调度量统一与工具可复核;工程更到位:明确的数据配比、训练日程与算力规模,保证性能与可复现性。更重要的是,这不是“做题型”模型的又一次包装:它以206B 级多学科科学数据为基础,覆盖科学文本、纯科学序列(DNA/RNA/蛋白/SMILES)、科学序列—文本/科学序列—科学序列成对数据,并在此之上统一建模、统一标注、统一度量,让“读—思—设”闭环可以被数据真实地驱动与校核。
数据集:https://huggingface.co/SciReason 模型:https://huggingface.co/SciReason 代码:https://github.com/open-sciencelab/SciReason
一、这是什么:一个科学数据“读—思—设”专精的基座模型
这是一套统一的科学推理基座:在 206B 级跨学科科学语料与数据上预训练,随后经“三段式”对齐——大规模指令微调(≈4000 万样本)、退火式冷启动(诱导长链思维)、以及引入任务分组与连续化科学奖励的强化学习(DAPO)——把自然语言与异构科学表示(DNA/RNA/蛋白、分子 SMILES、材料结构与文本等)深度对齐,覆盖从文本↔科学格式互译、知识/要素抽取、性质预测/分类到序列生成与可约束设计在内的完整工作流,单模型覆盖 5 大能力,共103个子任务。
与“做题型模型”的区别:
题库式/考试式模型:以高等教育考题数据为基础,侧重少量文本问答与模板化推导,缺少多表示科学数据(序列、符号、结构)的一致编码与可逆互译,无法直达科学研究一线。 本模型SciReasoner:以“文本+纯序列+序列—文本/序列—序列配对”的AI-ready 科学数据做预训练母体;在后训练阶段统一 I/O 模式、统一标签域(如 <SMILES>、<dna>、<protein>),并以物理/化学一致性的专业工具校验奖励和连续化奖励作为优化目标,确保推理与设计可被数据与度量反向约束。


二、三大方法学亮点:让“该想的时候认真想”
1)自适应科学推理(Adaptive Scientific Reasoning)
模型区分“即时型(instant)”与“思考型(thinking)”两类任务:前者保留直接答案监督,后者用思维链数据全量替换,确保在真正需要多步推理时给出连贯可查的理由,而简单任务保持高效直答。进一步的实证结论:在任务级别进行“思维链替换”优于在同一任务内混配CoT与非CoT目标,避免风格/长度不一致带来的校准偏移。
2)任务分组奖励(Task Grouped Rewarding)
将科学任务划分为距离度量型(数值预测)、匹配度量型(检索/抽取)与专业工具校验型三类,分别设计可比的质量度量与优势估计,提升跨任务的泛化与鲁棒性。
3)科学奖励“软化”(Scientific Reward Softening)
把原本难以优化的0/1正确性信号,统一映射为[0,1]的连续奖励(如把BERTScore、RMSE等标准量规规范化),显著改善复杂科学推理任务的收敛性与性能提升。
三、能力版图与代表成绩:覆盖“全栈”科学智能
五大能力家族:①文本↔科学表示的双向翻译;②文本/知识抽取;③性质回归预测;④性质分类;⑤序列生成与设计——任务总量涵盖至103类。 纵向对比:在54项任务上拿到SOTA,并在101项任务里名列前二,显示统一骨干在跨学科迁移与端到端工作流覆盖上的优势。 典型任务覆盖:分子表征互译(SMILES↔IUPAC/Formula)、分子描述/图说、蛋白功能翻译、跨模态知识抽取到材料/生物/化学性质预测与分类,再到约束可控的序列与材料/分子设计,强调“可逆性、守恒性、可检验性”的科学语义保真。

四、能落地到哪些场景
药物与分子设计:以性质回归/分类为“筛”,可控序列/分子生成为“设”,工具校验与知识检索为“评”,构成“筛—设—评”的可迭代数据闭环,降低无效合成与实验成本。 蛋白/核酸工程:从功能/本体描述与信息抽取,到稳定性/可溶性等属性预测,再到定向序列设计,兼顾规则约束与可达性。 材料科学:围绕文本—结构—数值三证据链开展性质回归/分类与候选筛选,适配主流数据库字段与口径。 合作交流:欢迎联系唐诗翔(tangshixiang@pjlab.org.cn)、白磊(bailei@pjlab.org.cn)