将 ScienceAI 设为星标
第一时间掌握
新鲜的 AI for Science 资讯


编辑丨ScienceAI
复杂疾病就像一场错综复杂的谜局:基因与环境因素在体内交织,导致细胞状态千变万化,也让我们难以看透病程背后的真相,进而影响了新药的研发。虽然单细胞测序技术能为每个细胞「拍照留念」,现有的分析方式却往往只能看到零散的瞬间,无法还原疾病发展过程中细胞状态的连续变化。
更麻烦的是,现有的计算模型难以从这些数据中自动找出关键的基因调控网络,也无法精准锁定那些加剧病情的「元凶」基因和通路。与此同时,现有的用于模拟药物扰动的模型还高度依赖昂贵且耗时的单细胞扰动实验数据,难以在实际新药筛选中大规模应用。
正因为如此,我们迫切需要一种全新的、一站式的计算平台,既能对时间序列单细胞数据进行动态建模,揭示疾病演进机理,又能无监督地进行「虚拟药物扰动 」效果预测,填补这一领域的多重空白。
2025 年 6 月 20 日,由麦吉尔大学丁俊教授和耶鲁大学 Kaminski 教授领衔的团队,推出了专为处理时间序列单细胞转录组数据而设计的深度生成式神经网络 UNAGI。该工具能够精确捕捉疾病进程中的细胞动力学变化,构建虚拟细胞与疾病模型,并在隐空间中准确模拟药物扰动的作用。

以特发性肺纤维化(IPF)患者数据为例,UNAGI 学得的疾病特异性细胞低维隐空间表征,不仅加深了对病程进展的认识,还帮助筛选出潜在的治疗药物候选。蛋白质组学实验证实,UNAGI 在分析细胞动态方面具有极高准确性;在精准切割肺切片(PCLS)的人体肺纤维化模型实验中,硝苯地平(Nifedipine,一种常见的降压药)的抗纤维化效果与 UNAGI 的预测高度一致,验证了 UNAGI 在药物筛选中的可靠性和预测准确性。
此外,UNAGI 适用范围广泛,现已应用于一些复杂疾病 —— 一方面展示了其在解析复杂细胞动力学和虚拟药物筛选方面的灵活性,另一方面凸显了其在多种病理背景下发现治疗方案的潜力。
UNAGI 是一种无监督深度学习方法 —— 这是一种无需预先标注样本即可在海量数据中挖掘潜在模式的 AI 方法。它采用了基于迭代变分自动编码器 - 生成对抗网络(iterative VAE-GAN)深度学习架构,同时针对归一化后常见的多样化数据分布进行优化。
通过这种深度生成式方法,UNAGI 能将高维基因表达数据压缩为疾病特异性的低维隐空间表征,构建出「虚拟细胞」。在模型学习过程中,这些隐空间表征融合了疾病特异性基因和调控信号的特征,因此能够反映出在疾病过程中不同细胞亚型的差异和功能,为后续的虚拟疾病的建模和虚拟药物扰动的模拟奠定基础。
在得到每个「虚拟细胞」后, UNAGI 利用时间序列数据重建「虚拟疾病」模型。 UNAGI 首先分析单细胞转录组数据,追踪基因在每个细胞中的时序表达变化,以学习健康细胞向病态细胞状态的转化轨迹;具体来说,在获得细胞隐空间低维表达后,UNAGI 对不同疾病阶段的细胞进行聚类,将不同疾病分期下的细胞簇按时间顺序连接(反映疾病进程中细胞从健康到疾病的状态演变),并据此推断驱动这些转变的基因调控网络。
UNAGI 能从数据集中描绘出多条不同细胞类型的细胞状态转换轨迹,从而构建一张图谱,即「虚拟疾病」模型。该「虚拟疾病」模型,既可描绘疾病过程中的细胞类型变化,又可捕捉疾病相关标记和基因调控因子。同时 UNAGI 利用了迭代训练的方式让模型专注于学习疾病中的关键的基因调控因子,从而生成疾病特异性的「虚拟细胞」从而更好地构建「虚拟疾病」模型。
在「虚拟疾病」模型基础上,UNAGI 集成了体外药物扰动模块,支持虚拟扰动(in silico perturbations)的功能。虚拟扰动能够利用计算机模拟候选药物对于疾病的干预效果。现有的药物数据库,如 Connectivity Map(CMAP)数据库中提供了药物 - 靶点基因信息。UNAGI 利用这些药物 - 靶点信息针对疾病细胞进行虚拟扰动,衡量这些药物对于疾病细胞隐空间表征的影响,模拟药物作用后细胞状态向健康状态转变的程度,从而对药物疗效进行实证评估。同样的方法也可用于探索治疗通路,为高通量、交互式的药物筛选与机制研究提供一站式解决方案。

图 1:UNAGI 框架流程图
该研究将 UNAGI 应用于特发性肺纤维化(IPF)单细胞数据集,利用在隐空间中完成聚类与可视化对于生成的「虚拟细胞」进行分析(图 3a)。聚类结果显示 UNAGI 能够捕捉到诸如纤维化成纤维细胞和气道成纤维细胞等亚群。这些结果表明随着疾病进展,纤维化细胞不断增多。它还揭示了不同细胞群的异质性差异,比如成纤维细胞在肺泡成纤维细胞和外膜成纤维细胞中均表现出很强的异质性。
与对照组相比,特发性肺纤维化患者中成纤维细胞比例显著上升,从不足 15% 增长至超过 40%(图 3b),这符合成纤维细胞积聚是特发性肺纤维化进展的典型特征。其中,肺泡成纤维细胞的增幅最为显著,而纤维化成纤维细胞仅在后期纤维化分期中出现。同时,血管内皮细胞比例则随着特发性肺纤维化的进展持续下降。基于这些细胞嵌入,UNAGI 揭示了各纤维化分期中细胞群的动态演变,为构建描述疾病进展的时序动态图谱奠定了坚实基础。
该研究还将 UNAGI 与其他细胞隐空间表征生成方法进行比较(图 3c),实验结果说明 UNAGI 生成的隐空间细胞表征质量好于传统分析方法(scanpy,Seurat),现有的深度学习方法(scVI),和单细胞转录组大模型(scGPT,Geneformer)。
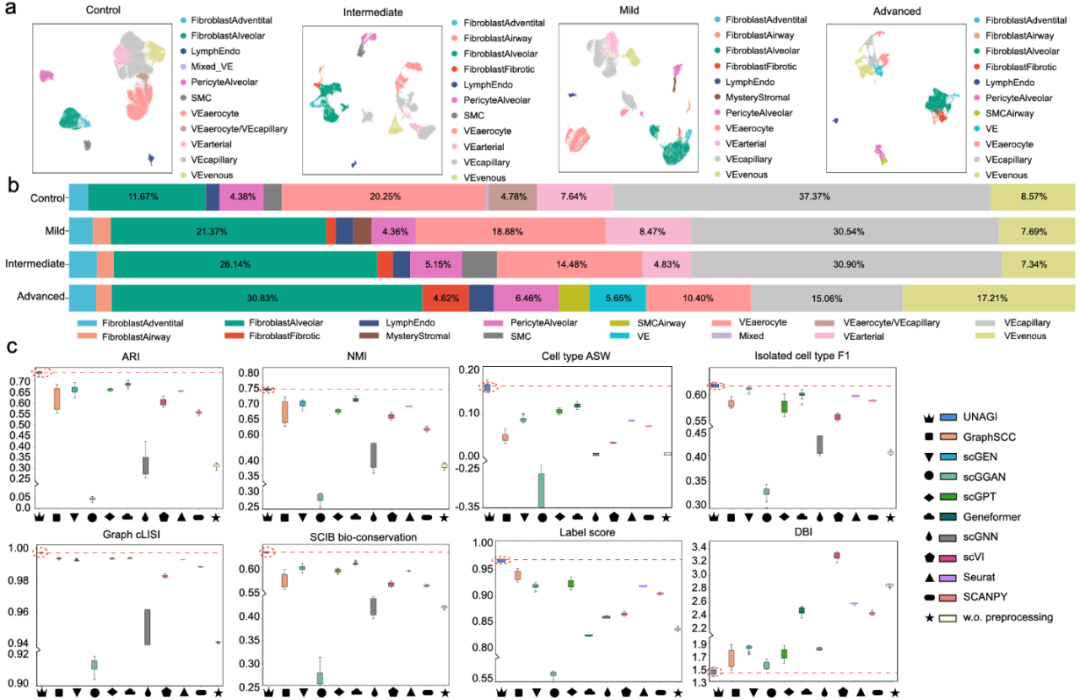
图 2:UNAGI对于疾病细胞隐空间的表达能力
UNAGI 建立的「虚拟特发性肺纤维化」模型共含有十条不同的疾病进展轨迹(图 4a),每条轨迹覆盖从健康到晚期纤维化阶段。其中,FibAlv-4 轨迹刻画了肺泡成纤维细胞在特发性肺纤维化进展过程中的状态演变(图 4b)。在这一轨迹对应的基因调控网络中,UNAGI 揭示了 CTCF、RAD21、SMC3 等关键调控因子,以及促纤维化因子 EP300 在 IPF 病程中的核心地位。富集分析结果不仅包含已知的胶原与细胞外基质通路,还发现了较为新颖的 SLIT/Robo 信号通路;此外,UNAGI 还捕捉到如 NCAM1 相互作用等尚未被确认为特发性肺纤维化驱动因子的潜在通路。
通过这种高深度基因调控网络重构,UNAGI 不仅锁定了核心调控因子和通路,还精准识别了它们的靶基因。特别是那些在不同纤维化分期中差异表达的靶基因,为重建特发性肺纤维化进程中的时序基因调控网络提供了关键线索。综上所述,UNAGI 对既有公认又相对未知的纤维化相关基因调控因子、信号通路及其靶基因的全谱式发掘,充分展现了 UNAGI 建立的「虚拟疾病」模型在揭示特发性肺纤维化进程中复杂分子交互机制方面的能力。
此外,该研究还对模型发现疾病相关的通路和基因标志物的能力进行了对比(图 4c,d),同样地 UNAGI 相比于传统分析方法(scanpy,Seurat),现有的深度学习方法(scVI)和单细胞大模型(scGPT,Geneformer),都更能发现疾病相关的生物信号。

图 3:UNAGI建立虚拟疾病模型并解析其背后的基因调控网络
该研究针对特发性肺纤维化进行了通路和药物层面的「虚拟扰动」实验,证明了 UNAGI 有能力无监督地寻找到潜在治疗靶点、通路和药物。针对特发性肺纤维化疾病的靶点通路,UNAGI 除了识别出经典特发性肺纤维化通路,如 TGFβ 通路和胶原生成通路,还发现了未被充分研究的新通路,比如 Netrin-1 信号通路和 GPCR 信号通路(图 5a)。在药物「虚拟扰动」方面,UNAGI 成功识别了 FDA 批准的特发性肺纤维化药物尼达尼布(nintedanib)和已完成 II 期临床试验的孤儿药酒石酸艾芬地尔(ifenprodil)(图 5b)。
除此之外,UNAGI 还预测了一些尚未与特发性肺纤维化建立明确关联却值得深入研究的高潜力候选药物,比如一种常用降压药:硝苯地平(Nifedipine)。耶鲁大学 Kaminski 博士团队通过精准切割肺切片(Precision-Cut Lung Slices)实验对这一预测进行了验证,结果显示硝苯地平能够显著降低组织中的纤维化标志物信号,证明了其潜在的治疗价值。在与其他能够进行「虚拟扰动」模型的对比中,UNAGI 的性能不但超过 scVI,scGEN 这类传统深度学习方法,也同样超过了单细胞大模型方法,包括 scGPT 和 Geneformer。

图 4:UNAGI发掘的潜在治疗IPF的通路和药物及UANGI的in-silico perturbation基准对比
在传统的 in vivo,in vitro 和 ex vivo 实验模型之外,该研究提出了一套「虚拟药物检验模型」框架,能够在单细胞层面高精度地模拟药物对细胞状态和基因调控网络的干预效果。该模型通过重构「虚拟细胞」与「虚拟疾病」过程,不仅加快了药物候选物的筛选速度,还大幅降低了实验成本与时间消耗。
借助 UNAGI 平台,研究人员可以快速且直观地评估千余种化合物对疾病进程的潜在影响,并优先确定最具疗效前景的靶点与药物组合。同时,这一方法与现有的动物和细胞实验互为补充,既可为传统的 in vivo,in vitro 和 ex vivo 研究提供假设与方向,也能在实验前阶段进行高通量、无监督的药效验证,从而显著提升新药研发的效率和成功率。
除 IPF 研究外,UNAGI 还已成功应用于杜氏肌营养不良症(Duchenne muscular dystrophy)等多种复杂疾病,彰显了其在 AI 驱动药物发现领域的广泛适用性。结合高分辨率单细胞组学,UNAGI 为揭示疾病机制、优先筛选治疗候选物,提供了一种可扩展、机制化且高性价比的解决方案。
此外,该研究成果还入选了《Nature》2024 年 11 月刊的科技专题「人类细胞图谱的计算技术」(Computational technologies of the Human Cell Atlas),该专题评述了全球七大领先工具,其中三项由丁俊博士及其团队开发,有效突破了长期制约单细胞研究进展的技术与资金瓶颈。
麦吉尔大学丁俊教授和耶鲁大学 Kaminski 教授为本论文的通讯作者。麦吉尔大学博士研究生郑昱旻和汉诺威医学院 Schupp 博士为本论文的共同第一作者。
论文地址:https://www.nature.com/articles/s41551-025-01423-7
人工智能 × [ 生物 神经科学 数学 物理 化学 材料 ]
「ScienceAI」关注人工智能与其他前沿技术及基础科学的交叉研究与融合发展。
欢迎关注标星,并点击右下角点赞和在看。
点击阅读原文,加入专业从业者社区,以获得更多交流合作机会及服务。










